Teoría de grafos y redes neuronales en la industria farmacéutica
La Cátedra iDanae (inteligencia, datos, análisis y estrategia) en big data y analytics, creada en el marco de colaboración de la Universidad Politécnica de Madrid (UPM) y Management Solutions, publica su newsletter trimestral correspondiente al 2T24 sobre sobre la teoría de grafos y redes neuronales en la industria farmacéutica
Teoría de grafos y redes neuronales en la industria farmacéutica
Introducción
En un mundo donde la tecnología y la ciencia avanzan de forma vertiginosa, la teoría de grafos se erige como una herramienta poderosa y versátil para su aplicación en el campo de la industria farmacéutica y la salud. Desde su origen en el siglo XVIII con los trabajos de Euler hasta su aplicación moderna en la bioinformática, los grafos han demostrado su capacidad para resolver problemas complejos y optimizar procesos en diversas áreas. Asimismo, se han desarrollado técnicas de inteligencia artificial basadas en grafos, como las redes complejas o las redes neuronales de grafos.
Hoy en día, estas técnicas pueden tener una aplicación relevante en la predicción de interacciones medicamentosas, el análisis de mutaciones genéticas y el reposicionamiento de fármacos, áreas críticas para el desarrollo de tratamientos más efectivos y seguros.
La combinación de avances en secuenciación genómica, inteligencia artificial y grandes volúmenes de datos ha permitido un mayor desarrollo en la práctica clínica; por ejemplo, optimizando terapias farmacológicas o mejorando la seguridad de los tratamientos. En los últimos años se han publicado diversos estudios en este sentido.
Por ejemplo, mCSM surge como una herramienta que utiliza firmas basadas en grafos para predecir los efectos de las mutaciones en proteínas. Aquí se demuestra que mCSM es eficaz para predecir cambios en la estabilidad de mutaciones en la proteína p53, superando a otros métodos y mostrando su utilidad en contextos de enfermedades complejas como el cáncer.
También se han utilizado redes neuronales convolucionales para extraer características locales y mecanismos de atención para obtener información importante en el campo de interacciones fármaco-proteína (DPIs).
En pruebas con datasets como C.elegans, humanos, y BindingDB, se demostró que el enfoque propuesto mejoraba la efectividad en la predicción de DPIs en comparación con métodos de aprendizaje automático convencionales. Cabe destacar que este tipo de estudios necesita alimentarse de buenas bases de datos, ámbito que también se ha desarrollado recientemente con datasets como DDInter [4]. Esta es una base de datos curada de interacciones entre fármacos (DDI) diseñada para ayudar a los médicos a identificar combinaciones de medicamentos peligrosas y mejorar los sistemas de salud. Además de las consultas básicas, incorpora una función de verificación de prescripción para ayudar a los médicos a decidir si las combinaciones de fármacos pueden utilizarse de forma segura.
En esta publicación se explora cómo la teoría de grafos y las redes neuronales (complejas y de grafos) están transformando la investigación farmacéutica. Se abordan los desafíos actuales en el desarrollo de medicamentos, como la predicción de la interacción medicamento-proteína y la interacción medicamento-medicamento, y cómo los métodos basados en estas técnicas de inteligencia artificial están proporcionando nuevas soluciones. Además, se presenta un caso de uso concreto de reposicionamiento de fármacos, destacando el potencial de estas tecnologías para acelerar la innovación y mejorar los resultados en salud.
Conexión entre los grafos y la industria farmacéutica
Panorama actual de la industria farmacéutica
En las últimas décadas, los avances significativos en la secuenciación del genoma humano y la bioinformática han catalizado una transformación sin precedentes en el ámbito de la medicina. Uno de los principales beneficiarios de este fenómeno es la industria farmacéutica, la cual ha emergido como un actor fundamental en el crecimiento y desarrollo de España. El crecimiento del Producto Interior Bruto (PIB) español hasta alcanzar un 3% en 2023, según datos del Instituto Nacional de Estadística (INE), es un claro indicador del impacto positivo que esta industria ha tenido en la economía del país. Este crecimiento económico se atribuye en gran medida a la capacidad de la industria farmacéutica para capitalizar los avances científicos y tecnológicos, así como para satisfacer las necesidades de salud de la población mediante la producción y comercialización de medicamentos innovadores.
En el ámbito de la investigación clínica de medicamentos innovadores, los laboratorios farmacéuticos nacionales desempeñan un papel destacado, con una inversión que supera los 750 millones de euros. España cuenta con una industria farmacéutica notablemente diversa, con más de 100 fabricantes de productos farmacéuticos básicos y más de 200 empresas especializadas en la preparación de medicamentos de alta complejidad. Además, importantes empresas farmacéuticas globales han establecido filiales en el territorio español.
El desarrollo de nuevos medicamentos en la industria farmacéutica, un proceso que puede tardar entre 10 y 15 años y requerir inversiones de hasta 800 millones de dólares, enfrenta desafíos considerables en términos de tiempo y recursos. Para acelerar este proceso, reducir costes y mejorar la efectividad de los fármacos existentes, se están integrando en la práctica clínica numerosos enfoques computacionales basados en inteligencia artificial y el uso masivo de datos. Estos enfoques permiten optimizar la terapia farmacológica, mejorando la seguridad y eficacia del tratamiento en beneficio de los pacientes. Dentro de la industria farmacéutica, se exploran numerosas líneas de investigación destinadas a lograr los objetivos mencionados, destacando tres enfoques principales: la predicción de la interacción medicamento-proteína, la predicción de mutaciones en aminoácidos y la predicción de interacción medicamento-medicamento. Esta convergencia entre la innovación tecnológica y la investigación biomédica promete revolucionar aún más el campo de la medicina, impulsando avances significativos para el tratamiento de enfermedades y mejorando la calidad de vida de las personas.
La teoría de grafos
La teoría de grafos es uno de los pilares fundamentales de las matemáticas discretas, cuyo origen se atribuye al famoso matemático suizo, Leonhard Euler. Él fue quien introdujo el concepto de grafos en su trabajo Solutio problematis ad geometriam situs pertinentis (1736), para resolver el problema de los siete puentes de Königsberg. Así se inició toda una nueva rama de las matemáticas, que siguió desarrollándose durante el siglo XIX y principios del siglo XX, gracias a las contribuciones de matemáticos como Arthur Cayley y Gustav Kirchhoff en el estudio de árboles y grafos conectados. En la segunda mitad del siglo XX, la teoría de grafos comenzó a aplicarse en informática, especialmente en algoritmos de redes, optimización y teoría de la computación. Esto se consolidó con trabajos como los de Dijkstra y su algoritmo para encontrar el camino más corto de un grafo. A partir de la década de 1990, y con la inclusión del estudio de redes de interacción reales, surgió la ciencia de redes complejas, que extiende la teoría de grafos al incorporar la dinámica y evolución de estos grafos en el tiempo. Una red compleja se refiere a una red, modelada como grafo, que posee ciertas propiedades estadísticas y topológicas no triviales. Las redes complejas de hoy en día se emplean en el estudio de fenómenos críticos de la física estadística, en la resolución de problemas bioinspirados o en las ciencias sociales, explorando cómo se comportan los sistemas bajo diferentes condiciones. Así, la teoría de grafos y las redes complejas coexisten de forma que la primera sirve como fundamento matemático y la segunda amplía su alcance para abordar problemas dinámicos y aplicados en el mundo real.
Un grafo es un objeto matemático que consiste en un conjunto de nodos (o vértices) y un conjunto de aristas (o enlaces) que conectan pares de nodos. Se representan mediante diagramas como el de la Fig. 1, de manera que los nodos se asocian con puntos y las aristas con líneas que unen nodos conectados entre sí. Permiten representar de manera simple estructuras de conectividad muy complejas. Es por esto que los grafos son esenciales en el estudio de redes complejas, ya que proporcionan la base matemática necesaria para modelar, entender y analizar el fenómeno de estudio.
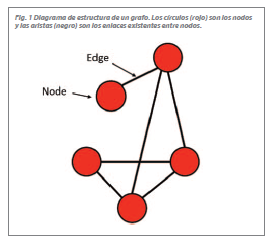
Es importante entender los conceptos de nodos y aristas. Los primeros son los elementos básicos de una red y representan las entidades individuales dentro del sistema. Su definición depende en gran medida de la red de estudio y su contexto. No es lo mismo estudiar el cerebro si se considera cada neurona como un nodo, o si se define cada nodo como una región cerebral. Las aristas, por otro lado, representan las interacciones o enlaces dados entre los nodos. Estas relaciones pueden ser físicas, como en redes de transporte, o abstractas, como en redes de amistad.
La teoría de grafos y las redes complejas se han convertido en herramientas con un gran potencial para entender y analizar sistemas en una amplia variedad de campos.
En ciencias sociales, el análisis de redes sociales permite comprender cómo se difunde la información, cómo se estructuran las comunidades y cuál es la influencia de ciertos individuos dentro de una red social. Esto se puede aplicar en política, para entender la dinámica de movimientos sociales y la propagación de ideas, o en el ámbito de las redes sociales digitales, para entender cómo se forman y evolucionan las conexiones entre usuarios, permitiendo mejorar los algoritmos de recomendación y personalización de contenido. Las redes complejas se han convertido en esenciales en diversos estudios de biología y medicina. Con ellas se pueden estudiar interacciones entre proteínas, redes neuronales o propagación de enfermedades. Estas últimas son cruciales para modelar la propagación de enfermedades, lo que permite anticiparse y diseñar estrategias de intervención más efectivas, como las campañas de vacunación o las medidas de cuarentena. Las redes tecnológicas son otro ejemplo. Incluyen internet y redes de transporte. Estas redes ayudan a optimizar la eficiencia y la robustez de estos sistemas frente a fallos. La estructura y funcionamiento de la red global de Internet, por ejemplo, se basa en principios de grafos y redes complejas.
En el ámbito económico y financiero, la teoría de redes proporciona herramientas para modelar las complejas interacciones en los mercados financieros y las transacciones económicas. Esto permite a los economistas y analistas financieros identificar posibles puntos de riesgo sistémico y desarrollar estrategias para mitigar el impacto de crisis financieras.
Por último, en ecología, se utilizan para modelar las interacciones dentro de ecosistemas, como las redes alimentarias. Esto ayuda a entender la estabilidad de los ecosistemas y el cómo diferentes especies pueden depender unas de otras, lo que permite detectar especies clave para la preservación del medio natural.
Un denominador común en todos estos campos es la idea de que las redes complejas permiten entender las interacciones y la dinámica subyacentes del caso de estudio. Mientras que en otros ámbitos de modelización es necesario recurrir a técnicas de feature engineering para comprender la relación entre variables, las redes de grafos llevan inherente esa relación por cómo está concebida su construcción misma. En un principio, cada nodo alberga la información y características que lo definen exclusivamente a él, pero la propia estructura de la red ya predetermina cuáles y cómo van a ser las diferentes interacciones con su entorno, de modo que, en última instancia, los vectores de características de cada nodo pasarán a incorporar la información relativa a su entorno y a cómo interacciona con sus vecinos. En resumen, la teoría de grafos y las redes complejas no solo ofrecen una forma de representar y analizar estructuras y sistemas complejos, sino que también proporcionan conocimientos cruciales para predecir comportamientos y diseñar intervenciones efectivas en una variedad de campos.
Redes complejas y redes neuronales de grafos
La evolución de la teoría de grafos ha dado lugar al desarrollo de redes complejas y redes neuronales de grafos, herramientas que permiten modelar y analizar sistemas dinámicos y en diversos campos, incluyendo la investigación farmacéutica. Estas redes proporcionan un marco robusto para entender las interacciones complejas entre distintos elementos de un sistema, como moléculas, proteínas y medicamentos, a través del uso de métodos avanzados de inteligencia artificial.
Redes complejas
Las redes complejas son grafos caracterizados por patrones de conexión no triviales que reflejan las propiedades y relaciones inherentes a los sistemas reales. A diferencia de los grafos simples, las redes complejas incluyen características como la modularidad, la robustez y la capacidad de adaptación a través del tiempo. Estas propiedades son esenciales para modelar sistemas biológicos y químicos donde las interacciones no son estáticas ni homogéneas:
- Estructura y dinámica. Las redes complejas integran tanto la topología (estructura estática) como la dinámica (comportamiento en el tiempo) de los sistemas. En el contexto farmacéutico, esto permite modelar cómo los medicamentos interactúan con múltiples proteínas y cómo estas interacciones pueden cambiar debido a mutaciones o la presencia de otras moléculas.
- Aplicaciones en biomedicina. Las aplicaciones de redes complejas en la biomedicina incluyen la identificación de nuevas relaciones entre genes y enfermedades, la modelización de la propagación de enfermedades infecciosas y la optimización de terapias combinadas. Estos modelos ayudan a predecir efectos secundarios y a personalizar tratamientos médicos.
Redes neuronales de grafos
Las redes neuronales de grafos (GNN, por sus siglas en inglés) representan una evolución de los métodos de aprendizaje automático que pueden operar directamente sobre estructuras de grafos. Las GNN son capaces de aprender representaciones de los nodos y las aristas que reflejan las características y las relaciones de estos elementos en el grafo.
- Capacidades de las GNN. Las redes neuronales de grafos son particularmente efectivas para tareas donde las relaciones estructurales entre datos son cruciales. Por ejemplo, en la predicción de interacciones medicamento-proteína, las GNN pueden integrar datos de estructura molecular y bioactividad para hacer predicciones precisas sobre cómo un fármaco interactuará con una proteína objetivo.
- Integración con datos biomédicos. La combinación de GNN con bases de datos biomédicas permite la extracción de características complejas y la identificación de patrones no evidentes a simple vista. Esto es útil para la predicción de efectos de mutaciones, donde las GNN pueden modelar cómo una alteración en la secuencia de aminoácidos afecta la estructura y función de una proteína.
Aplicaciones en la investigación farmacéutica
En las últimas décadas, las redes complejas han emergido como una potente herramienta de modelización. Destaca su aplicación en procesos de percolación, sincronización, epidemiológicos o transiciones de fase, entre otros. El desarrollo que las redes complejas han experimentado en estos campos ha permitido, también, que se expanda su ámbito de aplicación, llegando a utilizarse en la modelización y análisis de interacciones entre moléculas, proteínas y enfermedades, lo cual ofrece nuevas perspectivas y soluciones innovadoras para el descubrimiento de fármacos y el diseño de medicamentos. A continuación, se exponen algunas de las propuestas más prometedoras en este campo.
Predicción medicamento-proteína
Cuando un fármaco (una molécula química) se une a un objetivo biológico (como las proteínas), modula su comportamiento con el objetivo de devolverlo a su estado normal. Predecir esta interacción entre fármacos y objetivos (Drug–protein interaction, DPI) es una etapa muy importante en el proceso de descubrimiento de nuevos medicamentos y la comprensión de sus efectos secundarios. Un ejemplo claro de la importancia de investigar las interacciones fármaco-proteína es el surgimiento de la pandemia de COVID-19. Ante la ausencia de medicamentos específicos para combatir esta enfermedad, la investigación detallada de las interacciones entre las proteínas del virus SARS-CoV-2 y diversos fármacos existentes permitió identificar compuestos con potencial terapéutico. El estudio detallado de la proteína espiga del SARS-CoV-2, así como de otras proteínas virales clave como la proteasa 3CLpro, permitió desarrollar terapias antivirales y guiar el diseño de nuevos inhibidores específicos.
Dentro de este campo de estudio, han surgido las redes de interacción medicamento-proteína, redes orientadas a representar las interacciones entre fármacos y proteínas objetivo en el cuerpo humano. Los nodos se corresponden con los fármacos y proteínas, y las aristas representan las diferentes interacciones entre ellos. El desarrollo de estas redes puede ayudar a predecir la eficacia y los efectos secundarios de los fármacos, así como a identificar posibles combinaciones de fármacos para nuevas terapias.
La mayoría de los enfoques se basan en la información sobre fármacos y proteínas representada por vectores de características. Por ejemplo, uno de los enfoques comunes que suele formularse es el de obtener predicciones de DTI como una tarea de clasificación binaria. Estos enfoques hacen uso de bases de datos como BindingDB, que contiene información sobre las interacciones de unión entre proteínas y ligandos, que son moléculas pequeñas como los fármacos. Su objetivo principal es proporcionar datos experimentales detallados sobre la afinidad de unión, que es una medida de cuán fuertemente un ligando se une a una proteína. KIBA (Kinase Inhibitor BioActivity) es una colección de datos diseñada para facilitar la investigación en el ámbito del descubrimiento de fármacos, particularmente en relación con los inhibidores de quinasas. KIBA integra información de varias bases de datos de bioactividad y estandariza los datos de interacción entre quinasas y sus inhibidores.
Propiedades de los grafosLos grafos presentan una serie de propiedades que permiten caracterizar y estudiar el comportamiento de redes en diversas aplicaciones prácticas y teóricas. Las principales propiedades de los grafos incluyen:
En función de las propiedades y características de la red en estudio, y de cómo quedan definidos los nodos y aristas de la misma, se pueden distinguir diferentes tipos de redes, a saber:
Estáticas o dinámicas: si la estructura de la red varía con el paso del tiempo.
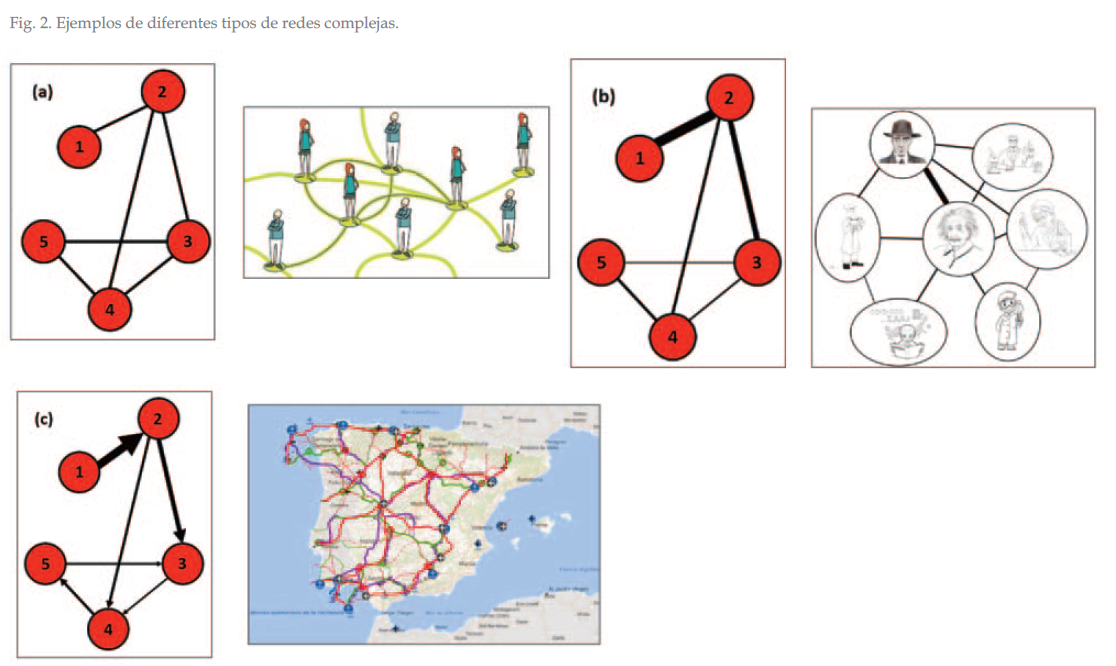 (a) Red de amigos en la escuela. Es una red no dirigida -ser amigos es una relación bidireccional- y no pesada -todas las relaciones de amistad tienen el mismo valor-. Puede ser no conexa si hay gente en clase que no tiene ningún amigo. Es homogénea y dinámica -las relaciones interpersonales varían con el tiempo-. Normalmente no sigue una ley de potencias, luego no es libre de escala. |
Predicción mutaciones en aminoácidos
La secuenciación del genoma humano ha revelado una gran diversidad genética en las poblaciones humanas, incluyendo mutaciones que alteran la secuencia de aminoácidos en las proteínas. Estas mutaciones concretas, llamadas variantes sin sentido, pueden ser patógenas, afectando la función de las proteínas y la salud de los pacientes, o benignas, con efectos mínimos. Se ha conseguido catalogar más de 15 millones de variaciones de nucleótidos en la población humana, sin embargo, muchas variantes aún se desconocen y muchas otras aún tienen un impacto desconocido, representando un desafío en la genética humana. El virus de la inmunodeficiencia humana (VIH), responsable de 630.000 muertes en 2022, presenta una alta tasa de mutaciones en sus proteínas. Los fármacos antirretrovirales aprobados para combatir el VIH están diseñados para inhibir dos proteínas específicas. Sin embargo, las mutaciones en los aminoácidos de estas proteínas pueden alterar su estructura, haciendo que los fármacos pierdan su efectividad y dejen de actuar contra el virus.
Los métodos computacionales se están usando para estudiar el mecanismo molecular de la resistencia a los medicamentos inducida por mutaciones y para desarrollar herramientas predictivas para detectar mutaciones que afectan a la secuencia de aminoácidos y, por tanto, a la estructura y función de la proteína. Un enfoque que ya ha obtenido buenos resultados es el del método mCSM (matriz de escaneo de corte de mutaciones), que utiliza firmas basadas en grafos para representar el entorno estructural de tipo salvaje y el aprendizaje automático para predecir el efecto de las mutaciones en la estabilidad de las proteínas. En este contexto, las proteínas se representan como redes donde los nodos son átomos y las aristas son las interacciones entre estos átomos. Al analizar cómo las mutaciones afectan las propiedades locales y globales de estas redes (por ejemplo, cambios en la conectividad o en la distribución de las interacciones), mCSM puede predecir el impacto de las mutaciones en la estabilidad y función de las proteínas. En Cambridge se han desarrollado extensiones del método mCSM para predecir el impacto de la mutación en proteína-ligando (mCSM‐lig) e interacciones proteína-proteína (mCSM‐PPI2), y más recientemente en Fiocruz, Brasil y en Melbourne, Australia, por Douglas Pires y David Ascher para proteína-ácido nucleico (mCSM‐NA), antígeno anticuerpo (mCSM‐AB) interacciones y conformaciones y dinámicas de proteínas en combinación basadas en la dinámica de modo normal (DynaMut).
Para poder desarrollar esta línea de investigación existen diversas bases de datos; entre las más destacadas se encuentran: UniProtKB alberga una extensa colección de información detallada y curada sobre proteínas, y proporciona datos sobre secuencias, funciones, estructuras tridimensionales, localización celular, interacciones con otras moléculas y anotaciones biológicas; DbSNP se centra en las variaciones genéticas, incluyendo mutaciones y polimorfismos de nucleótido único (SNPs); Human Gene Mutation Database contiene información detallada sobre mutaciones genéticas humanas, e incluye datos sobre la ubicación exacta de la mutación en el genoma, la naturaleza de la mutación (por ejemplo, si es una sustitución de un solo nucleótido, una inserción o una deleción), y cualquier efecto conocido de la mutación en la función génica o en la salud humana.
Predicción interacción medicamento-medicamento
La combinación de dos o más fármacos se conoce como terapia combinada, y es una estrategia común para mejorar la eficacia terapéutica y reducir los efectos secundarios. Sin embargo, una selección inadecuada de medicamentos puede resultar en reacciones adversas. Por ello, es de especial interés conocer las interacciones entre medicamentos (IDD) [36]. Los efectos que producen las IDD son un factor de riesgo importante para la hospitalización, especialmente entre pacientes ambulatorios de edad avanzada. De hecho, se estima que las IDD contribuyen del 5% al 14% de las reacciones adversas en pacientes hospitalizados. Un ejemplo de esta IDD peligrosa podría ser la terapia combinada de Warfarina (anticoagulante que previene formación de trombos) con antiinflamatorios no esteroideos (reducen inflamación, dolor y fiebre) como el ibuprofeno, que pueden causar hemorragia al inhibir el metabolismo del anticoagulante.
En esta línea de investigación se pueden emplear redes de interacción medicamento-medicamento. La idea subyacente es similar a las anteriores, con la particularidad de que los nodos representan fármacos y enfermedades, y las aristas indican la asociación entre ellos. En este caso, la intención es la de buscar posibles efectos secundarios y, toda vez localizados esos efectos secundarios, y entendido por qué surgen, conocer cómo prevenirlos o encontrar una posible solución. Los algoritmos predictivos aprovechan datos farmacocinéticos y farmacodinámicos para predecir cómo los medicamentos interactuarán en el cuerpo humano, así pueden identificar patrones y correlaciones que quizás no sean evidentes para los métodos tradicionales.
Los datos utilizados en estudios como el mencionado anteriormente se extraen de grandes bases de datos de interacciones conocidas como DrugBank y DDInter. DrugBank cuenta con un total de 570.091 productos farmacéuticos donde se incluyen medicamentos de molécula pequeña aprobados y medicamentos experimentales, entre otros. Proporciona más de 200 campos de datos para cada medicamento, con la mitad de la información dedicada a aspectos químicos, farmacológicos, farmacéuticos y otros aspectos del medicamento, y la otra mitad dedicada a documentar la secuencia, estructura y ruta del medicamento objetivo. DDInter contiene más de 236.834 IDD que involucran 1.833 medicamentos y documenta la información detallada sobre cada IDD, como mecanismos, niveles de riesgo, recomendaciones para el ajuste de medicamentos, etc.
Caso de uso: reposicionamiento de fármacos
El reposicionamiento de medicamentos consiste en identificar nuevos usos terapéuticos para medicamentos que previamente han sido aprobados para diferentes propósitos médicos. Hasta ahora, estos estudios dependían de exámenes manuales y técnicas estadísticas, métodos que requerían considerable tiempo y esfuerzo. Sin embargo, los nuevos métodos computacionales permiten analizar grandes volúmenes de datos genéticos y clínicos, descubriendo nuevos objetivos farmacológicos y prediciendo la eficacia y toxicidad de los compuestos con alta precisión; acelerando así el proceso de desarrollo de fármacos.
Algoritmos de Deep Learning, como los basados en redes neuronales de grafos, se utilizan para el estudio de reposicionamiento de fármacos. Estas herramientas han sido utilizadas para identificar fármacos inhibidores de Janus Kinase 2 (JAK2). La alteración de la función JAK se asocia con diversos trastornos inflamatorios, como la artritis reumatoide y la psoriasis, entre otras. La inhibición de esta enzima reduce las consecuencias negativas de estas enfermedades.
En este caso de uso concreto se han utilizado los datos procedentes de la base de datos DUD-E, que contiene propiedades fisicoquímicas de distintos medicamentos ya aprobados por la FDA. Estos datos han sido utilizados para el entrenamiento de una red convolucional gráfica (GCN) proporcionada por DeepChem, una biblioteca Python de código abierto que cuenta con el modelo GraphConvMol. Con este modelo se pretende determinar, con base en la estructura molecular, la capacidad inhibitoria de los medicamentos.
Este modelo de grafos convierte cada átomo del medicamento en un nodo y cada enlace covalente en una arista, de tal manera que cada átomo comunica sus características únicas a los átomos adyacentes. El conjunto de datos JAK2 se dividió en conjuntos de entrenamiento, validación y prueba en una proporción de 8:1:1 y luego se sometió al modelo GraphConvMol usando validación cruzada con 5 iteraciones.
El modelo entrenado, utilizando el algoritmo GraphConvMol de DeepChem, procesó medicamentos aprobados por la Administración de Alimentos y Medicamentos del Gobierno de los Estados Unidos (FDA) para evaluar su potencial de actividad inhibidora de JAK2. Se han identificado 20 compuestos activos que presentan actividad inhibitoria de JAK2, entre los cuales se encuentran algunos ya conocidos. La actividad inhibidora de JAK2 de los 20 fármacos detectados se evaluó experimentalmente y todos ellos mostraron una inhibición de la actividad enzimática JAK2.
Mediante la integración de GCN con técnicas de acoplamiento molecular y su aplicación a una base de datos de compuestos activos, se ha logrado una detección más exhaustiva de posibles inhibidores de JAK2. Este enfoque ha permitido identificar de manera eficiente nuevos candidatos a fármacos que no habían sido considerados anteriormente. Entre estos compuestos, se encuentran ribociclib, amodiaquina, topiroxostat y gefitinib, los cuales han demostrado un potencial inhibidor prometedor de JAK2. Además, la validación experimental ha corroborado los resultados obtenidos mediante aprendizaje profundo y acoplamiento molecular. Por consiguiente, se propone este procedimiento para el reposicionamiento de fármacos en un amplio espectro de objetivos terapéuticos.
Conclusiones
La teoría de grafos y su evolución a través de técnicas de inteligencia artificial han demostrado ser una herramienta con un gran potencial en la industria farmacéutica, abordando con éxito desafíos complejos en la predicción de interacciones medicamentosas, análisis de mutaciones genéticas y reposicionamiento de fármacos. Su integración con tecnologías avanzadas y el análisis de grandes volúmenes de datos ha optimizado significativamente la investigación y el desarrollo de medicamentos.
Los estudios recientes han validado la eficacia de métodos basados en grafos para predecir los efectos de mutaciones en proteínas y mejorar la estabilidad de mutaciones complejas. Asimismo, las redes neuronales convolucionales y otros enfoques de aprendizaje automático han mejorado la predicción de interacciones fármaco-proteína y fármaco-fármaco, cruciales para el desarrollo de terapias seguras y efectivas.
Además, la creación de bases de datos ha facilitado la identificación de combinaciones de medicamentos peligrosas y la optimización de las terapias farmacológicas. Estos avances subrayan la importancia de una colaboración continua entre la bioinformática, la teoría de grafos y la industria farmacéutica para mejorar los resultados en salud y acelerar la innovación.
En resumen, la teoría de grafos no solo ha revolucionado el campo de la investigación farmacéutica, sino que también ha abierto nuevas oportunidades para el desarrollo de tratamientos más efectivos y seguros. Con su capacidad para modelar y analizar sistemas complejos, los grafos seguirán desempeñando un papel crucial en la evolución de la medicina y la salud pública.
La newsletter ya está disponible para su descarga en la web de la Cátedra tanto en español como en inglés.